picrust2功能预测-从qiime2安装到数据分析_picrust2功能预测结果怎么看-程序员宅基地
技术标签: github linux picrust2 windows
记录一下跌跌撞撞地摸索过程
picrust2功能预测
参照公众号《宏基因组》刘永鑫的原创:《PICRUSt2:OTU或ASV等16S随便预测宏基因组且数据库增加10倍》https://blog.csdn.net/woodcorpse/article/details/89302863
注:命令中 \为断行用,实际上输入命令要去掉,在输入文件路径时没有用此符号断行。
虚拟机与qiime2安装
参照公众号《宏基因组》刘永鑫的原创:《QIIME 2用户文档. 01简介和安装 Introduction & Install(2020.2) 》使用VirtualBox方式安装部分
https://blog.csdn.net/woodcorpse/article/details/75103929
下载最新版本虚拟机:https://download.virtualbox.org/virtualbox/6.1.4/VirtualBox-6.1.4-136177-Win.exe
下载最新版本虚拟机扩展包:
https://download.virtualbox.org/virtualbox/6.1.4/Oracle_VM_VirtualBox_Extension_Pack-6.1.4.vbox-extpack
下载QIIME2镜像:https://data.qiime2.org/distro/core/2020.2
安装:
1) 安装最新版虚拟机,默认位置,装好后将扩展包拷贝到安装目录windows/program files/oracle/virtualbox,双击安装扩展包。
2) 双击下载好的QIIME2镜像或在虚拟机界面点击导入镜像,修改分配给虚拟机的cpu和内存(一般是windows的一半),点击导入。
3) 启动虚拟机,菜单中安装Guest Additions,按照提示完成后,重启linux系统才能设置共享文件夹和共享复制粘贴。https://mp.weixin.qq.com/s/WS9u0nhiS1eizL5KXKs__A?
4) 设置共享文件夹,添加共享文件夹,勾选固定分配/自动挂载。
问题1:安装好后,输入qiime的命令均显示command not found。
前几天看到有人在百度上发起了同样问题。不是安装有问题,是qiime的命令和qiime2的命令不兼容,因此使用qiime2进行16分析的命令可参照https://www.cnblogs.com/afeiyuanda/p/11037287.html
Picrust2安装
https://github.com/picrust/picrust2/官网有给出两种安装方法,建议第一种安装,不会出错。
安装好后第二次使用picrust,也要输入激活命令:
conda activate picrust2
或者
conda activate picrust
如果想要使用qiime2命令,则要先退出picrust2,输入失活命令:
conda deactivate
问题2:v2.3.2-b版本优化了匹配度,测序序列进行后续分析时都会出现这样的问题,stopping - all 234 input sequences aligned poorly to reference sequence(--min_align option specified a minimum proportion of 0.8 aligning to reference sequences
先反向互补完,再做功能预测就可以了,seqtk seq -r otus.fa.format.fasta > otus_rc.fa
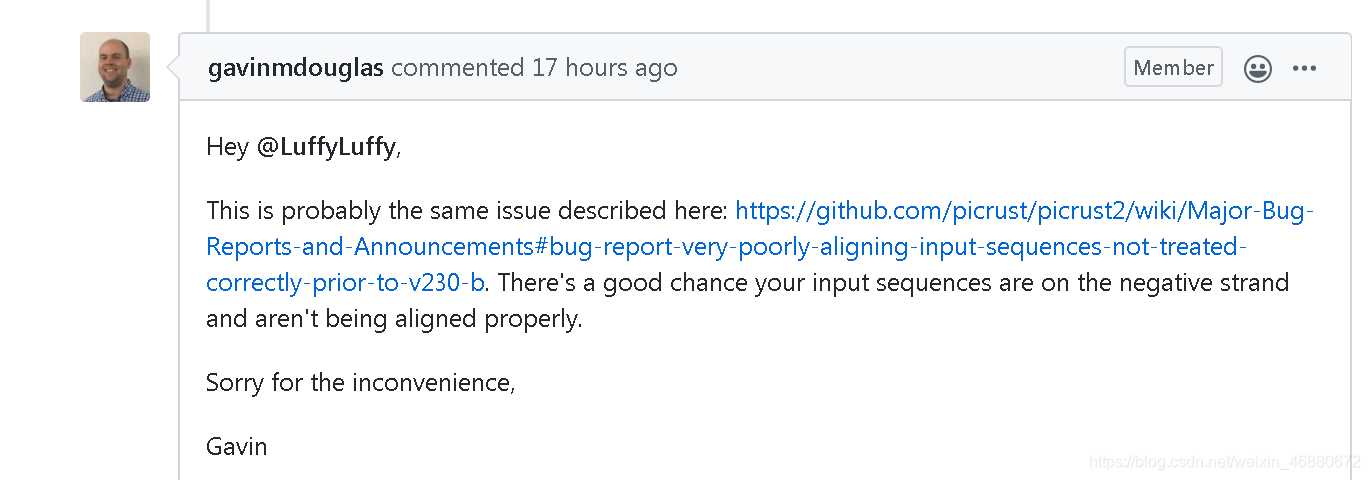
如果使用以前的版本不会出现这样的问题。
有个小插曲:不知道怎么解决这个问题的时候,无论输入什么数据都显示出这个结果,心态差点就崩了,然后花了点钱请淘宝上做生信分析的人,用我的数据帮我跑了一遍,可以出结果。最后观察版本之间有什么差别的时候就发现最新版本优化了,然后淘宝上那个生信技术厉害的客服用最新版本也出不了结果,他就去官网发了一个issue(就是上面图片里的),开发者很快就给回复了,解释了原因。
问题3:使用vsearch跑出的结果使用out.fa和out_table.biom两个文件可以直接跑出预测结果,但使用qiime跑出的结果中两个文件夹的out名称不一致,要删掉后面的其他字符可采用以下方法。
修改fastq文件OTU 名字`
awk '{print $1 }' FASTA_IN > FASTA_OUT
其中FASTA_IN和FASTA_OUT是输入和输出FASTA文件的名称。
$2:一行一行的读取指定的文件, 以空格作为分隔符,打印第二个字段
比如有这样一个文件
a1 b1 c1 d1
a2 b2 c2 d2
执行的结果是,输出:
b1
b2
问题4:会出现以下报错,大概率是电脑内存不够,picrust2官网有一个针对这个错误的issue。
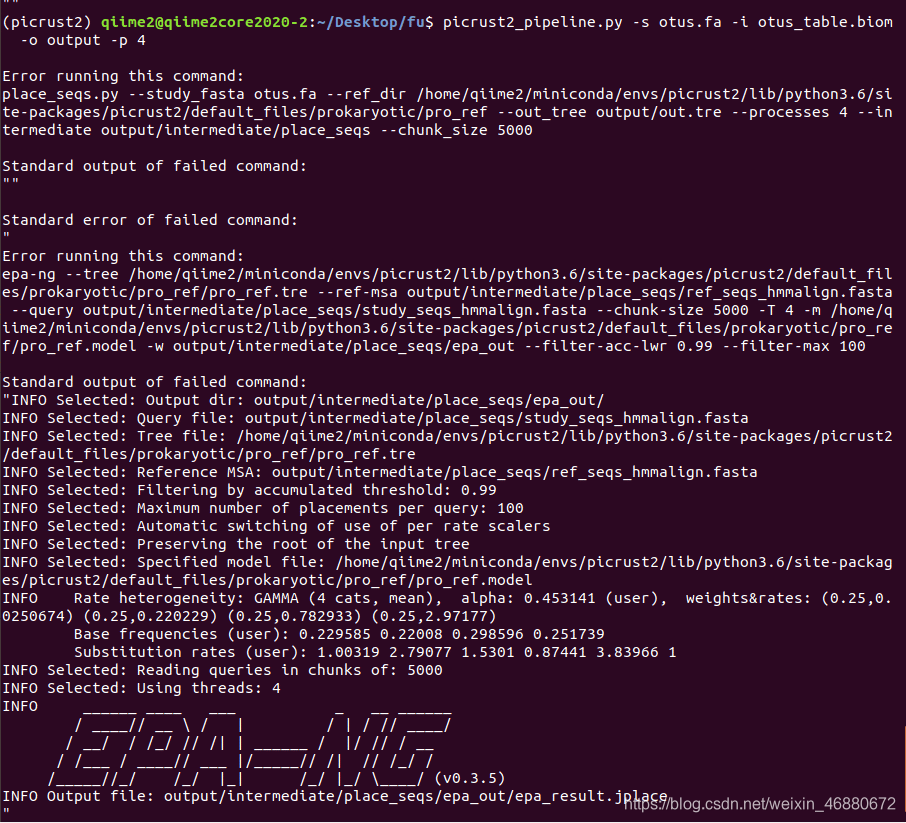
解决的根本办法是扩大你的内存,建议32G往上走,windows32G内存一般能够给虚拟机分配20G左右运行内存,在样本量比较多的情况下,往往16G是不够的,但是有时候样本量可以的情况下可以打擦边球试运行,降低CPU线程。我之前运行过分配给虚拟机36G的内存(windows总共48G)运行104个样本,使用4和2线程均无法得到结果,之后再使用1个线程耗时19000秒就成功了。也有试过88个样本,2线程9000秒。
###picrust2预测
比较完整的一步脚本是
picrust2_pipeline.py -s dna-sequences.fasta -i feature-table.biom -o \
picrust2_out_pipeline -p 20 -r default_files/prokaryotic/pro_ref/pro_ref \
--in_traits COG,EC,KO,PFAM,TIGRFAM
可能default_files/prokaryotic/pro_ref/pro_ref这个文件需要你在你的虚拟机上搜索后输入完整并且正确的文件路径。
这个命令运行后的结果包括COG,EC,KO,PFAM,TIGRFAM的丰度信息和MetaCyc途径丰度。
我一般输入的命令是:
picrust2_pipeline.py –s out.fa -i out_table.biom \
-o picrust2_out_pipeline -p 4
-p 4是使用4线程的意思
输出的结果包括EC,KO,PATHWAY的丰度信息。
但是但是结果文件中的结果比较简洁,比如只输入EC1.1.1,而不标注对应的酶名字,这时候需要加工加工。根据需要酌情添加描述:
add_descriptions.py -i EC_metagenome_out/pred_metagenome_unstrat.tsv \
-m EC -o EC_metagenome_out/pred_metagenome_unstrat_descrip.tsv
add_descriptions.py -i KO_metagenome_out/pred_metagenome_unstrat.tsv \
-m KO -o KO_metagenome_out/pred_metagenome_unstrat_descrip.tsv
add_descriptions.py -i COG_metagenome_out/pred_metagenome_unstrat.tsv \
-m COG -o COG_metagenome_out/pred_metagenome_unstrat_descrip.tsv
add_descriptions.py -i PFAM_metagenome_out/pred_metagenome_unstrat.tsv \
-m PFAM -o PFAM_metagenome_out/pred_metagenome_unstrat_descrip.tsv
add_descriptions.py -i TIGRFAM_metagenome_out/pred_metagenome_unstrat.tsv\
-m TIGRFAM -o TIGRFAM_metagenome_out/pred_metagenome_unstrat_descrip.tsv
add_descriptions.py -i pathways_out/path_abun_unstrat.tsv
-m METACYC -o pathways_out/path_abun_unstrat_descrip.tsv
结果输出中没有KEGG结果,可通过以下命令生成KEGG pathway 丰度表和注释结果
pathway_pipeline.py -i KO_metagenome_out/pred_metagenome_unstrat.tsv \
-o KEGG_pathways_out --no_regroup --map \
/home/qiime2/miniconda/envs/picrust/lib/python3.6/site-package/
picrust2/default_files/pathway_mapfiles/KEGG_pathways_to_KO.tsv
这里KEGG_pathways_to_KO.tsv需要你在你的虚拟机上搜索后输入完整并且正确的文件路径。
# 添加功能描述
add_descriptions.py -i KEGG_pathways_out/path_abun_unstrat.tsv.gz \
--custom_map_table /home/qiime2/miniconda/envs/picrust/lib/python3.6/site-
package/picrust2/default_files/description_mapfiles/KEGG_pathways_info.tsv.gz \
-o KEGG_pathways_out/path_abun_unstrat_descrip.tsv.gz
这里/KEGG_pathways_info.tsv.gz需要你在你的虚拟机上搜索后输入完整并且正确的文件路径。
如果不懂可看原文https://www.jianshu.com/p/e3beaee77423
或如果不想一步生成结果,就看官网分步命令:https://github.com/picrust/picrust2/wiki/PICRUSt2-Tutorial-(v2.1.4-beta)
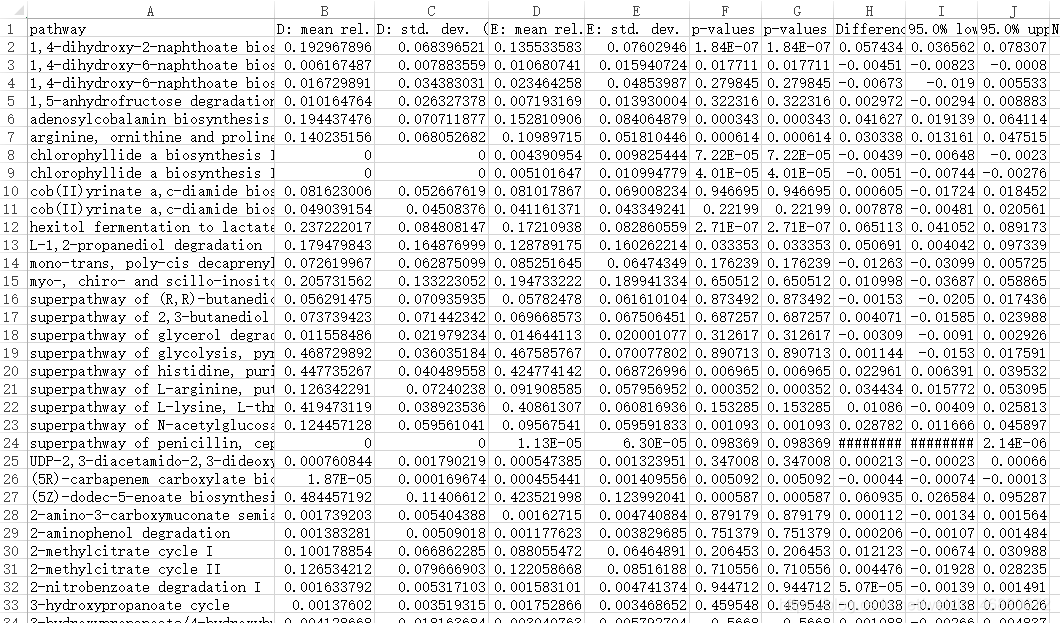
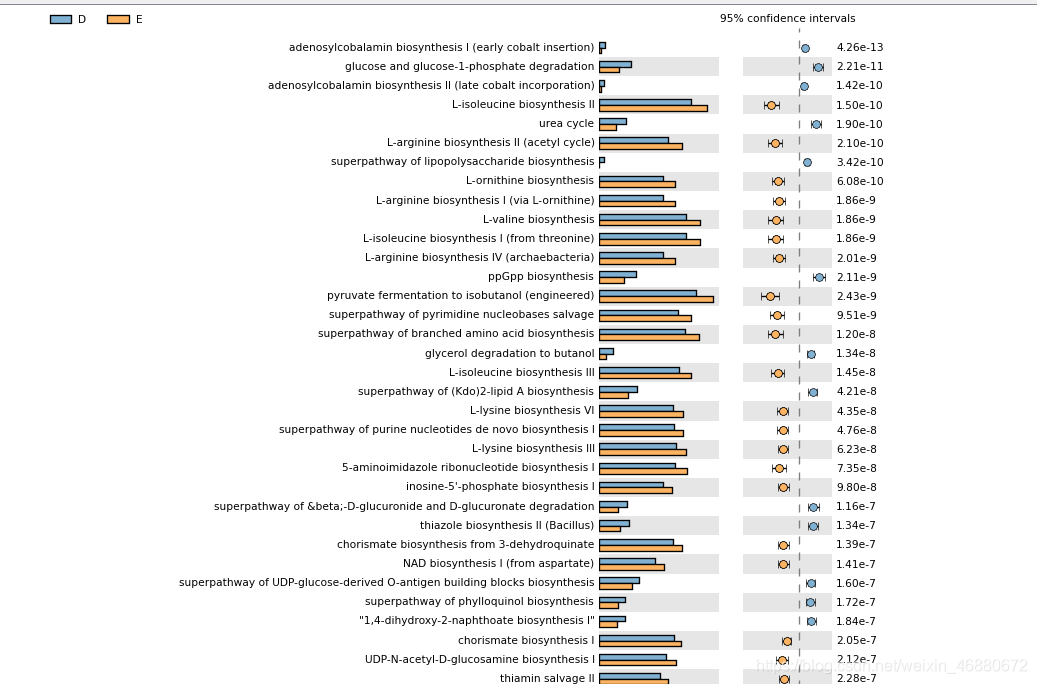
分析好的结果,使用STAMP软件进行显著性差异分析,该软件导入相应数据后既可以直接出很直观漂亮的显著性差异图,也可一次性导出所有通路或酶的组间差异分析详细数据。如下两图所示。
智能推荐
计算机的外围设备简介_计算机外围固定-程序员宅基地
文章浏览阅读6.1k次,点赞3次,收藏5次。外围设备介绍计算机的外围设备(简称外设)虽然很多,但按功能分大类只有四类:输入、输出、存储、网络通讯。有些专业计算机需要的外围设备也不尽相同,并不都需要这四类外围设备。外围设备可以按需要组装,有些专业计算机甚至可以将存储设备和主芯片集成到一片芯片上,从而不再需要外加存储设备。最早的计算机(那时还只能称为计算器,只能做简单运算,如ABC机和ENIAC机)输入只是一些拨码开关,只能输入数字(还得是二进_计算机外围固定
java 图片中加文字_java怎么在图片上加文字-程序员宅基地
文章浏览阅读1.5k次。java 图片中加文字_java怎么在图片上加文字
GBase8cGDCA认证模拟题题库(三)_如果需要打开delete语句的审计功能,需要开启下面哪个参数-程序员宅基地
文章浏览阅读720次,点赞20次,收藏6次。B 选项,在创建模式时,可以不指定模式名。C 选项,兼容模式可选值为 AB、C、PG.安装GBase 8c分布式集群时所需的配置文件gbase.yml,在解压GBase8cV5 S3.0.0BXX CentOS x86 64.tar.bz2压缩包生成的目录中得到。真值的有效文本值是: TRUE、t、"true'、y、yes'、"1'TRUE'、true、整数范围内1~2^63-1、整数范围内-1~-2^63。GBase 8c 使用create table 创建表时,不指定参数,默认是astore,行存表。_如果需要打开delete语句的审计功能,需要开启下面哪个参数
xml文件中几个名词_xml文件里面的名词-程序员宅基地
文章浏览阅读334次。1 xmlns是XML Namespaces的缩写,中文名称是XML(标准通用标记语言的子集)命名空间。 web-app是web.xml的根节点标签名称 version是版本的意思 xmlns是web.xml文件用到的命名空间 xmlns:xsi是指web.xml遵守xml规范 xsi:schemaLocation是指具体用到的schema资源_xml文件里面的名词
【OpenGL】中点圆、椭圆生成算法_用setpixel函数中点画圆算法代码c++-程序员宅基地
文章浏览阅读1.6w次,点赞12次,收藏69次。OpenGL 中点圆、椭圆生成算法_用setpixel函数中点画圆算法代码c++
HTML-CSS实现背景图片出现不同的位置_css背景图高度占据一半另一半有别的背景色-程序员宅基地
文章浏览阅读2.1k次。首先在HTML中写入div,命名为img,在这个div中加入一个span标签并命名为img-bg和img50(5星为50).<div class="img"> <span class="img-bg img50"></span> <span class="img-bg img45"></span> <span class="img-bg img40"></span> </div> 在css代码._css背景图高度占据一半另一半有别的背景色
随便推点
matlab建模DNA双链,PPT绘制科研图形—DNA双链、分子细胞模型-程序员宅基地
文章浏览阅读1.3k次。原标题:PPT绘制科研图形—DNA双链、分子细胞模型 PPT绘制DNA双链 1用矩形工具画一个矩形如下,线条颜色设置为无,填充色如下图蓝色 2选中矩形框,选择菜单栏的“格式—— 编辑形状——转换为任意多边形” 3这个时候再看下“编辑形状”,可以看到“编辑顶点” 已经为可用状态 4点击“编辑顶点“,矩形框四个角变为黑色实点。可以拖动实点变为如下图示。然后在边缘上右键,选择”添加顶点“,添加如下顶点 ..._matlab双螺旋结构模型图怎么画
duilib vs2015 安装_DuiLib(1)——简单的win32窗口-程序员宅基地
文章浏览阅读169次。资源下载https://yunpan.cn/cqF6icWRN5CTc 访问密码 92e3 注:DUILIB库.7z 是vs2015下编译好的动态库及静态库,如上图所示一、新建一个win32工程项目设置中选择:debug,常规中:全程无优化-全程无优化,多线程调试 (/MTd);我的项目选择的是静态编译,使用的是静态库,就不需要带duilib.dll文件了代码如下:#include #inclu..._vs2015使用duilib
OpenGL: 渲染管线理论详解_通过此次实验你对固定渲染管线的opengl编程有什么了解。-程序员宅基地
文章浏览阅读5k次,点赞4次,收藏13次。学习着色器,并理解着色器的工作机制,就要对OpenGL的固定功能管线有深入的了解。首先要知道几个OpenGL的术语:渲染(rendering):计算机根据模型(model)创建图像的过程。模型(model):根据几何图元创建的物体(object)。几何图元:包括点、直线和多边形等,它是通过顶点(vertex)指定的。 最终完成了渲染的图像是由在屏幕上绘制的像素组成的。在内存中,和像素有关的信息(如像素的颜色)组织成位平面的形式,位平面是一块内存区域,保存了屏幕上每个像素的一个位的信息。_通过此次实验你对固定渲染管线的opengl编程有什么了解。
Android MPAndroidChart:动态添加统计数据线【8】_android 动态统计-程序员宅基地
文章浏览阅读3.9k次。Android MPAndroidChart:动态添加统计数据线【8】本文在附录相关文章6的基础上,动态的依次增加若干条统计折线(相当于批量增加数据点)。布局文件:
vmware中的linux虚拟机如何增加磁盘容量_linux虚拟机磁盘空间不足-程序员宅基地
文章浏览阅读6.3k次。vmware中 centos的磁盘大小 20G->30G现象:fdisk -l可以看到增大后的磁盘总量,但是需要增加分区并格式化然后挂载才能使用.一、vmware中的设置先关闭虚拟机vm->settings->hard disk->utilities->expand->输入大小(增加后的大小)二、启动虚拟机,进入命令行1、 fdisk /dev/sda进入命令行Comman_linux虚拟机磁盘空间不足
Hadoop2.7.3下Mysql8.0下Hive2.3.8的安装_hive2.3.8安装-程序员宅基地
文章浏览阅读927次。hive安装前提:1.基于hadoop2.7的完全分布式集群搭建完成hadoop2.7集群搭建2.MySQL8.0安装完成 安装centos7上MySQL8.0Hive2.3.8的安装下载链接:https://mirrors.tuna.tsinghua.edu.cn/apache/下滑找到hive点击进去点击hive2.3.9(hive2.3.9和hive2.3.8差别不大)下载画红线的也就是bin.tar.gz后缀的hive解压安装下载完成后通过xftp传到虚拟机上(基操不在赘述)_hive2.3.8安装